利用CRISPR/Cas9技术创制Rc基因恢复红稻
时间:2023-06-11 09:35:22 来源:雅意学习网 本文已影响 人 
张元野 尹丽颖 李荣田,* 何明良 刘欣欣 潘婷婷 田晓杰 卜庆云 李秀峰,*
利用CRISPR/Cas9技术创制基因恢复红稻
张元野1尹丽颖1李荣田1,*何明良2刘欣欣3潘婷婷4田晓杰2卜庆云2李秀峰2,*
(1黑龙江大学 生命科学学院, 哈尔滨 150080;
2中国科学院 东北地理与农业生态研究所, 哈尔滨 150081;3东北林业大学 生命科学学院,哈尔滨 150040;
4黑龙江八一农垦大学 农学院, 黑龙江 大庆 163319;*通信联系人, email: lirongtian07@aliyun.com; lixiufeng@iga.ac.cn)
【目的】将栽培稻品种恢复为米质优、抗逆性强的红稻具有较大的研究价值。利用CRISPR/Cas9基因编辑技术,编辑原花青素转录调节因子基因,恢复红种皮特性,以改良水稻米质,提升抗逆性。【方法】利用CRISPR/Cas9技术,以为靶基因,构建突变载体pYLCRISPR/Cas9--gRNA,以空育180、上育453为材料,转化获得转基因植株,通过测序手段和表型观察验证成果。【结果】分子水平检测获得突变材料2种,其中KY-1在1414―1417 bp缺失4个碱基,终止子突变为苯丙氨酸;
SY-1在1411 bp处缺失1个碱基,终止子突变为天冬氨酸。2种编辑材料均恢复为红米表型,且具有一定耐盐碱能力。【结论】利用CRISPR/Cas9基因编辑技术成功获得恢复红种皮表型的纯合株系,为红米改良提供基础材料。
水稻;
;
红种皮;
耐盐碱;
CRISPR/Cas9
随着我国国民生活水平的不断提高,人们关于健康饮食方面的意识不断增强[1]。作为主要粮食作物的水稻,民众的关注度不再满足于其产量,对品质的要求逐步提高,特种稻米的市场需求增加[2]。特种稻米分为色稻、香稻和专用稻。红米作为有色稻米的一种,属于特种稻米[3]。我国拥有较为丰富的有色稻米种质,占水稻资源总体的10%,其中红米种质有8963份[4]。有色稻米相较于普通稻米(白米),蕴含丰富的微量元素和营养物质,拥有广泛的市场[5],因而挖掘和创制红米种质尤为重要。
世界上绝大部分种植水稻种皮颜色为白色[6],主要由和两对等位基因控制稻米红色种皮性状[7]。在驯化过程中,调控水稻种皮颜色的原花青素转录因子基因,在第6外显子后出现14 bp的碱基缺失[8],造成移码突变,提前出现终止子,使得后续的bHLH结构域缺失,基因不能完整表达,种皮为白色;
当与基因同时存在时,种皮为红色;
当基因单独存在时,表现为棕色或棕色伴随深色斑点;
当基因单独存在时,种皮表现为白色[9]。在世界范围内,杂草稻是稻田系统中难以根除的一种竞争性杂草,形态特征介于野生稻和栽培稻之间[10],大多数杂草稻种皮颜色为红色[11],具有生长周期短、谷壳颜色深、耐胁迫能力强[12]等特点,因而在恢复红米表型后,有望恢复部分杂草稻的耐胁迫能力,如耐盐碱能力。
现今,CRISPR/Cas9技术作为一种非常成熟的基因编辑技术,因其简单高效被广泛应用于植物分子育种当中[13],在水稻[14]、玉米[15]、大豆[16]等粮食作物中均有应用,特别是在水稻中,研究者采用该技术对粒型[17]、香味[18]、直链淀粉含量[19]等多个性状进行了改良,为进一步推广特种稻米的优良种质奠定了基础。目前,传统育种仍是红米培育的主要方式,红稻8号[20]、清红优1号[21]等具有优良农艺性状的红米品种,均为通过传统育种方式培育而成。随着分子技术的革新,研究者已将调控红米性状的基因逐步定位[22],并加以克隆,Zhu等[23]通过CRISPR/Cas9技术成功恢复了白米材料的红米表型。因而,本研究试图应用CRISPR/Cas9基因编辑技术,将抗逆性强、产量高、耐倒伏的优质水稻恢复红色种皮。
本研究选择日本优质早粳稻上育453和空育180(均为空育131的姊妹系)作为试验材料,这两个品种具有圆粒型、结实率高、抗倒伏及耐寒性强等优质特性,适宜北方寒地种植,改变其米色具有较大的应用推广前景。利用CRISPR/Cas9技术,在基因终止子前进行基因编辑,定向突变基因第6外显子1407 bp附近的碱基序列,使得该位点附近碱基缺失,产生移码突变,剔除终止子,恢复原始氨基酸序列,将第474位终止子变为苯丙氨酸或天冬氨酸,bHLH结构域得以正常表达,使其表现出红米性状,耐胁迫能力提高,为深入开展红米品种改良工作奠定材料基础。
1.1 试验材料
本研究以适宜寒地种植的日本优质早粳稻上育453和空育180为试验材料,获得基因编辑材料。所有材料在哈尔滨的室外,以25 cm×25 cm的间距种植于盆栽盒,自然长日照条件下生长。
pYL-U6a-gRNA和pYLCRISPR/Cas9Pubi-H双元载体[24]由华南农业大学刘耀光院士惠赠。
1.2 CRISPR/Cas9靶点接头设计及载体构建
通过NCBI(https://www.ncbi.nlm.nih.gov/)获得(LOC_Os07g11020)的基因序列,利用CRISPR-GE在线网站(http://skl.scau.edu.cn/),在第6外显子(图1)设计2对靶点接头引物CAS9-Rc1/2-LP/RP(表1),利用CRISPR Primer Designer软件,进行水稻全基因组序列比对,检测靶点特异性,排除潜在脱靶序列。
参照Ma等[25]的方法进行载体构建,将连接好的载体通过热激法转入大肠杆菌Match1-T1中,菌液PCR检测后,保存菌液。把菌液送至库美生物有限公司,利用测序公共引物M13-F测序,测序成功后将返还的质粒通过热激法转入EHA105农杆菌感受态细胞中。
1.3 T0代阳性植株的获得
通过农杆菌介导法,将构建好的表达载体转入日本优质早粳稻上育453、空育180的愈伤组织中,经过潮霉素筛选抗性愈伤组织,分化获得T0植株。将T0植株种植于室外盆栽盒中,对每株T0代植株提取叶片的全基因组DNA,通过引物HPT-F/R(表1)进行PCR检测,产物大小为900 bp的植株为阳性转基因植株,并收获种子用于下一代种植。
1.4 T1代纯合突变植株的筛选
在T0代收获的种子中,挑选粒型饱满且剥壳后种皮为红色的种子,室温下浸种1 d,37℃下催芽2 d,育秧后种植于室外盆栽盒中。在苗期提取T1代植株基因组DNA,利用测序引物Rc-F/R(序列列于表1)进行PCR扩增,扩增产物送去库美生物科技有限公司进行测序,选择测序成功的植株收获种子继续加代。
1.5 T2代植株耐盐碱能力分析
依据测序结果,选择红种皮种子分别进行芽期耐盐碱实验。选择颗粒饱满的种子剥皮后,室温浸种1 d,37℃下催芽2 d,选取出芽一致的种子播种于装有发芽纸的培养皿中,分别加0、30、100、200 mmol/L NaCl溶液,每2~3 d测量一次芽长,挑选表型差异较大处理的植株,使用试剂盒提取叶片RNA,根据前人研究选择3个上调的耐盐标记基因([26]、[27]、[28],表1),qRT-PCR鉴定表达量。另选取发芽一致的种子播种于装有发芽纸的平皿中,分别加0、20、40 mmol/L Na2CO3溶液,每2~3 d测量一次芽长,挑选表型差异较大处理的植株,使用试剂盒提取叶片RNA,根据前人研究选择3个上调的耐碱标记基因([29]、[30]、[31],表1),qRT-PCR鉴定表达量。同时选取正常生长、20 mmol/L Na2CO3处理、200 mmol/L NaCl处理下的各组材料RNA,以qRT-PCR鉴定表达量(表1)。

黑色序列为靶点序列,灰色下划线序列为PAM序列。
Fig. 1. Gene structure and target site of

表1 本研究中所用的引物
2.1 T0代转基因阳性苗检测
白种皮水稻基因编码区序列全长为1483 bp,编码473个氨基酸,根据软件预测,在终止子前25 bp内,寻找合适PAM位点,利用CRISPR/Cas9技术敲除部分碱基,产生移码突变,去除终止子,恢复bHLH结构域,即恢复完整基因功能。据此设计CRISPR/Cas9靶点引物(图1),构建Cas9-Rc载体,利用农杆菌转化法侵染上育453、空育180的愈伤组织,获得转基因植株T0代,对获得的14株转基因植株进行潮霉素鉴定,其中13株为阳性植株,1株为阴性植株,阳性率为92.85%(图2),将阳性植株种植于室外的盆栽盒中,收取种子。
2.2 T1代植株表型及突变类型鉴定
将T0代收获的种子剥壳处理,观察是否恢复红米表型。在收获的13个株系中,有2个株系的种皮表型变为红色,其中空育180背景有1株,命名为KY-1,上育453背景有1株,命名为SY-1,可明显观察到红米表型变化(图3-A)。
测序发现,KY-1在1414―1417 bp处缺失4个碱基(CCAA),导致第472位脯氨酸(Pro)、第473位精氨酸(Arg)、第474位终止子分别突变为甘氨酸(Gly)、天冬氨酸(Asp)、苯丙氨酸(Phe);
SY-1在1411 bp处缺失1个碱基(C),导致第472位脯氨酸(Pro)、第473位精氨酸(Arg)、第474位终止子分别突变为谷氨酰胺(Gln)、甘氨酸(Gly)、天冬氨酸(Asp)(图3)。相较于正常表达的基因,KY-1、SY-1在bHLH结构域前缺失多个氨基酸,其中KY-1在471-476位缺失6个氨基酸,且存在T470C、Q477H的突变;
SY-1在471―475位缺失5个氨基酸,且存在T470C的突变,但二者bHLH结构域均无变化(图3-D),能够正常表达。两种类型的突变均可以使得白米氨基酸序列第474位终止子提前解除,恢复bHLH结构域的表达及红米的表型。
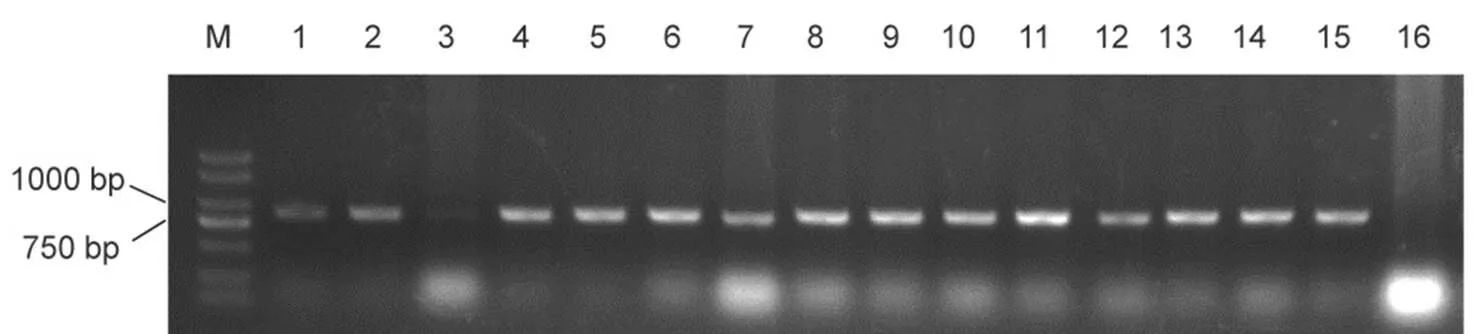
M―DM2000 DNA 标记; 1~14―T0代植株; 15―阳性对照; 16―阴性对照。
Fig. 2. Transgenic detection of T0generation plants.

A―红米性状恢复株系与野生型表型对比(标尺=1 cm);
B―红米性状恢复株系与野生型碱基序列比对;
C―红米性状恢复株系与野生型氨基酸序列比对; D―红米性状恢复株系与野生型bHLH结构域比对(红框内为bHLH结构域)。
Fig. 3. Phenotype and sequencing identification of T1transgenic plants.
2.3 T2代植株抗性分析
用0 mmol/L NaCl处理,红米株系SY-1于4 d后表现出较强的生长能力;
在其他浓度下,0~4 d内,SY-1并未表现出突出的生长能力,而后逐渐超过野生型及其他株系(图4-A~D)。在200 mmol/L NaCl处理下,SY-1相较于其他材料表现出较强的耐盐能力(图4-D)。以空育180为背景的KY-1,仅在30 mmol/L NaCl处理下,表现出一定的耐盐碱能力(图4-B)。
在SY-1中,三种标记基因、、的表达水平,与野生型相比均有显著提高(图5-A~C),与表型差异相符(图4-G);
而在KY-1中,、的表达水平相较于野生型无明显差异,仅的表达量有所升高(图5),与其基因编辑株系的弱耐盐能力表型一致(图4-G)。
在20 mmol/LNa2CO3处理下,7 d后红米株系SY-1表现出较为突出的生长能力,KY-1与野生型差异不大(图4-E);
在40 mmol/LNa2CO3的处理下,各株系虽表现出不同的生长趋势,但差异并不显著(图4-F),因而选择20 mmol/LNa2CO3处理下的野生型、KY-1、SY-1植株提取RNA进行表达量鉴定,发现三种耐碱标记基因、、的表达趋势各不相同,但相较于野生型,KY-1、SY-1的表达水平均有所升高(图5-D~F)。
同时选取正常生长、20 mmol/LNa2CO3处理、200 mmol/L NaCl处理下的各组材料测定表达量,在红米KY-1、SY-1中,不同处理下表达水平相较于白米(野生型)均有明显升高;
该差异在盐处理(图5-I)、碱处理(图5-H)下表现相似,在SY-1中,表达水平与其他材料差异显著,KY-1的表达水平有所提升。
本研究利用CRISPR/Cas9技术,以优质粳稻品种上育453、空育180为研究材料,对基因进行定点突变,通过产生移码突变剔除终止子,改变氨基酸序列,从分子手段上成功恢复红米表型,相比于传统的杂交培育优质红稻的方法,在技术和效率上有极大的提高,很大程度上缩短育种年限,成功获得优质红稻。
在驯化水稻过程中,绝大部分水稻的基因第6外显子区域存在14 bp的碱基缺失,导致氨基酸序列提前终止,使得红米表型丧失,研究发现,在的终止子前增减正确的碱基数目后,bHLH结构域能够正常表达,较产物虽缺失一段短肽,仍可恢复红米表型。红稻对非生物胁迫具有一定的耐受能力,杂草红稻能够在稻田中顽固生长,因而期望种皮性状恢复后,能够恢复部分非生物胁迫耐受能力。
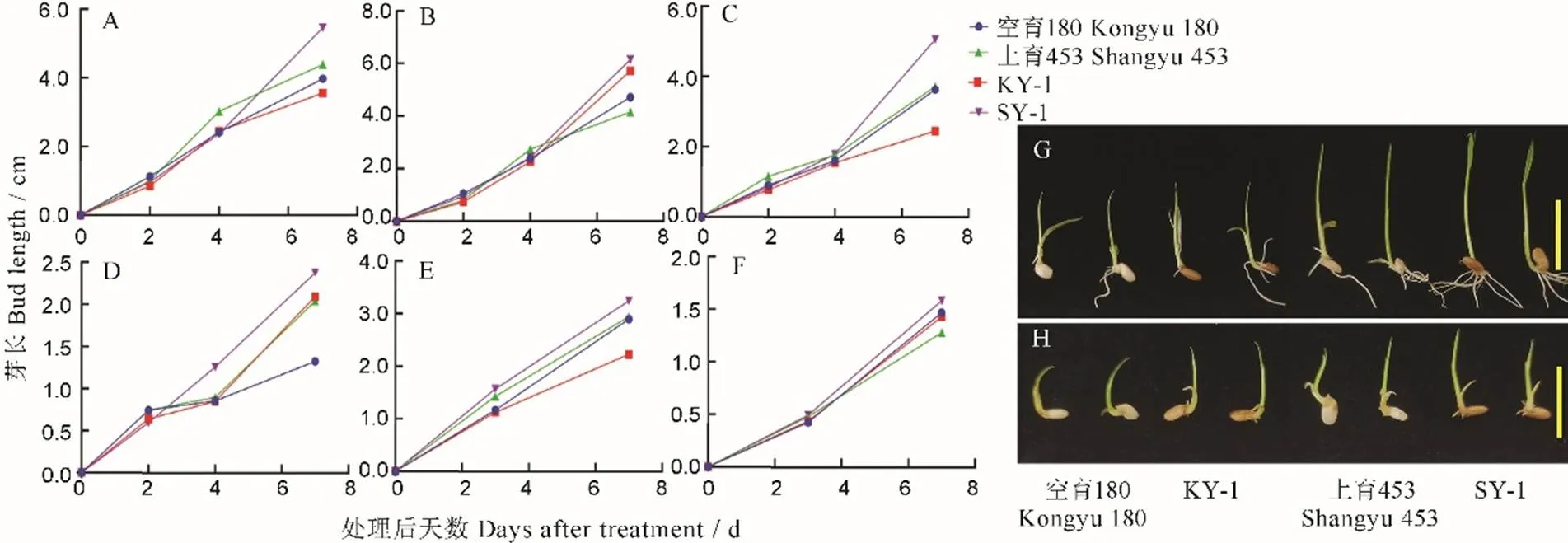
A~D分别为0、30、100、200 mmol/L NaCl溶液处理下各组的芽长;
E~F分别为20和10mmol/L Na2CO3下各组的芽长;
G―200 mmol/L NaCl处理的芽长(标尺=2 cm);
H―20 mmol/L Na2CO3处理的幼苗生长情况(标尺=2 cm)。
Fig. 4. Identification of salt and alkali tolerance of T2generation transgenic plants.
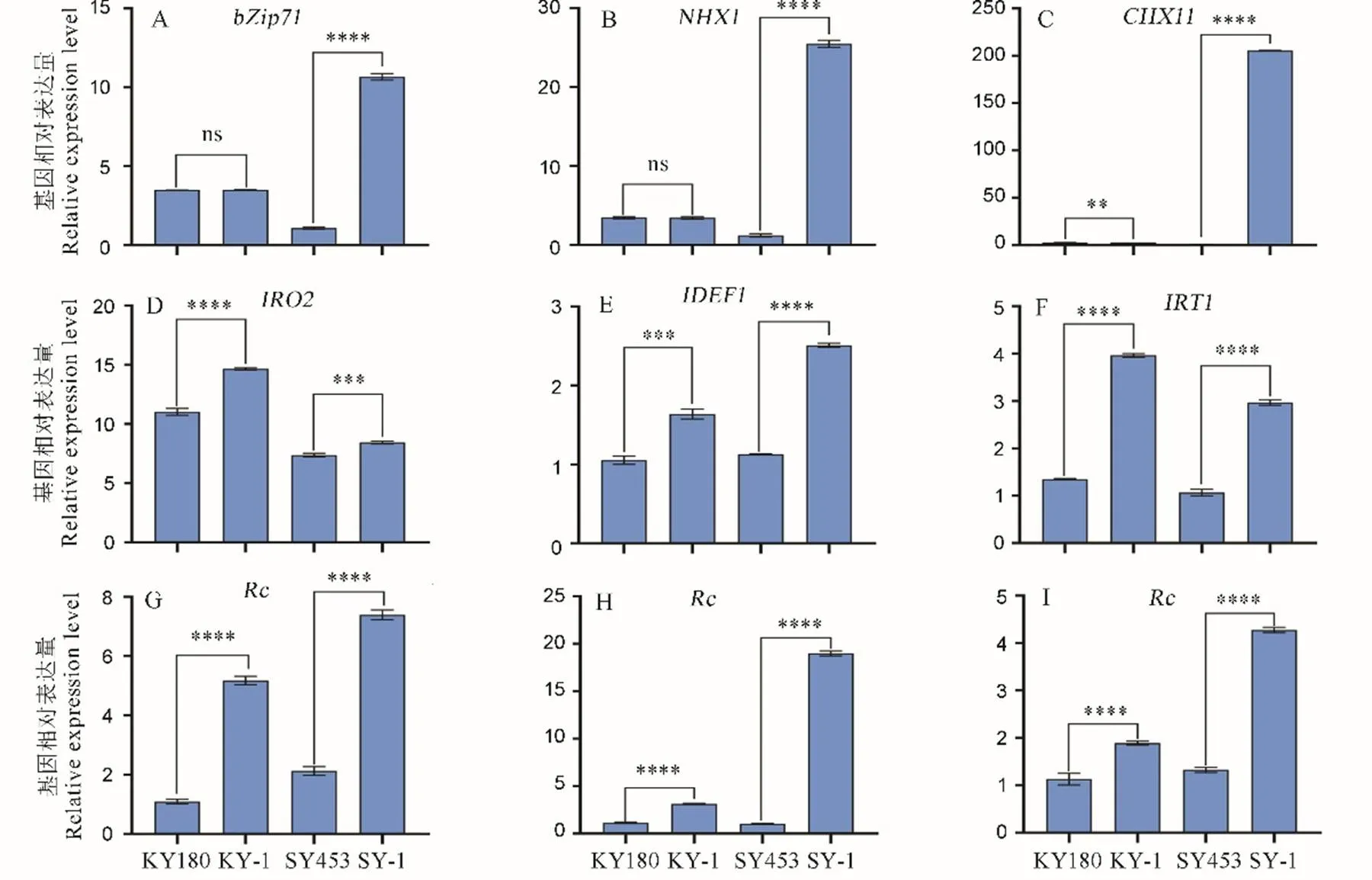
A~C―200 mmol/L NaCl处理下耐盐标记基因表达量;
D~F―20 mmol/L Na2CO3处理下耐碱标记基因表达量;
G~I―200 mmol/L NaCl、20 mmol/L Na2CO3及空白对照Rc表达量。ns表示无显著差异、**、***、****表示分别在0.005、0.001、0.0001水平上显著差异(t检验)。KY180―空育180;
SY453―上育453;
KY-1―空育180的基因编辑株系;
SY-1―上育453的基因编辑株系。
Fig. 5. Identification of saline-alkali tolerance related gene expression levels in T2transgenic plants.
根据前人研究[26-31],参与多条代谢通路调节根系细胞生长和能量转化,能提高非生物应激能力;
参与调控钠离子转运,参与调控钙离子转运,在盐应激反应中,具有调节渗透压提高盐胁迫耐受能力的作用,盐处理条件下,、、均上调表达,将其选作标记基因研究耐盐能力;
、、均为铁离子调节因子,能够维持植物体内铁稳态,耐受土壤pH变化保障植物正常生长,在碱处理条件下,均上调表达,将其选作标记基因研究耐碱能力。在对T2代植株耐盐碱试验中,SY-1表现出良好的耐盐碱能力,且标记基因表达水平有明显差异,但KY-1的耐盐标记基因表达水平并未表现出明显提高,而在耐碱试验中观察到有差异的表型,且耐碱标记基因表达水平上存在一定差异,证明恢复红种皮性状能够提高部分耐盐碱能力,导致KY-1与SY-1在耐盐能力上表现出的不同趋势的原因尚不清楚,推测可能与不同材料背景下的遗传差异有关,也可能与标记基因的选择有关,可进行进一步的研究。
在本研究中,虽获得转基因阳性植株13株,但剥皮发现一部分植株的种子并未表现出红米表型,即虽转化成功,但未产生有效的突变。在14 bp碱基缺失的基础上,增减正确数目的碱基后,方可继续表达缺失的bHLH结构域;
当插入过多或过少碱基时后续序列仍为无效的碱基序列,并不能正常表达bHLH结构域,红米表型无法有效恢复,而鉴定得到的两种编辑植株株系,均为纯合株系,性状遗传稳定,且基因表达水平相较于野生型(白米)有显著差异。虽期望恢复红米性状的编辑材料能够表现较强的耐胁迫能力,但耐盐碱的表型数据低于预期,而与本研究耐盐碱标记基因的联系尚不明朗,且基因表达量在正常生长、耐盐碱处理情况下均表现极显著差异,可在后续研究进一步探究编辑材料耐盐碱表型差异与基因在非生物胁迫中的作用。
目前,CRISPR/Cas9技术在植物研究领域广泛应用,尤其是在水稻的种质改良中。红米作为一种优质的特性,尚且存在很大的研究空间,虽在本研究中成功获得红米性状恢复株系,且效率远高于传统育种,依旧存在部分潮霉素筛选鉴定成功而恢复表型失败的株系,证明技术手段仍存在一定的不足;
且尚未在大田扩大规模种植,可在后续研究中进一步探究其他农艺性状是否存在差异。更优质的水稻种质培育,仍是科研工作者的远大目标,本研究可为优质水稻恢复红米性状提供一定的数据基础。
[1] 牛怡君. 基于大数据背景下健康管理的食品营养运用研究[J]. 现代食品, 2020(14): 129-131.
Niu Y J. Research on the application of food nutrition in health management based on big data[J]., 2020(14): 129-131. (in Chinese)
[2] 胡时开, 胡培松. 功能稻米研究现状与展望[J]. 中国水稻科学, 2021, 35(4): 311-325.
Hu S K, Hu P S. Research status and prospect of functional rice[J]., 2021, 35(4): 311-325. (in Chinese with English abstract)
[3] 李玉纯, 毕宝山, 郑美花. 特种稻米及开发前景[J]. 吉林农业, 2001(10): 10-11.
Li Y C, Bi B S, Zheng M H. Special rice and its development prospects[J]., 2001(10): 10-11. (in Chinese)
[4] 王子平. 中国红米资源的研究与利用进展[J]. 湖南农业科学, 2008, 36(4): 32-34.
Wang Z P. Research and utilization of red rice resources in China[J]., 2008, 36(4): 32-34. (in Chinese)
[5] Finocchiaro F, Ferrari B, Gianinetti A, Dall"Asta C, Pellegrini N. Characterization of antioxidant compounds of red and white rice and changes in total antioxidant capacity during processing[J]., 2010, 51(8): 1006-1019.
[6] 吴建, 余显权. 水稻红米色素基因的初步定位[J]. 山地农业生物学报, 2017, 36(3): 75-77.
Wu J, Yu X Q. Preliminary mapping of pigment genes in rice red rice[J]., 2017, 36(3): 75-77. (in Chinese with English abstract)
[7] Furukawa T, Maekawa M, Oki T, Suda I, Iida S, Shimada H, Takamure I, Kadowaki K. Theandgenes are involved in proanthocyanidin synthesis in rice pericarp[J]., 2007, 49(1): 91-102.
[8] Sweeney Megan T, Michael J T, Bernard E P, Susan M C. Caught Red-Handed:encodes a basic helix-loop-helix protein conditioning red pericarp in rice[J]., 2006, 18(2): 283-294.
[9] Sweeney M T, Thomson M J, Cho Y G, Park Y J, Williamson S H, Bustamante C D. Global dissemination of a single mutation conferring white pericarp in rice[J]., 2007, 3(8): e133.
[10] Wang W J, Zhao M H, Zhang G C, Liu Z M, Hua Y C, Jia X T, Song J Y, Ma D R, Sun J. Weedy rice as a novel gene resource: A genome-wide association study of anthocyanin biosynthesis and an evaluation of nutritional quality[J]., 2020, 11: 878.
[11] Juan C L, Sun L D, Ping S Z, Soo S H, Lu B R. Gene flow from cultivated rice () to its weedy and wild relatives[J]., 2004(1): 67.
[12] Lang H, He Y T, Zeng F L, Xu F, Zhao M, Ma D. Comparative transcriptomic and physiological analyses of weedy rice and cultivated rice to identify vital differentially expressed genes and pathways regulating the ABA response[J]., 2021, 11(1): 12881.
[13] Wang M, Mao Y, Lu Y, Wang Z, Tao X, Zhu, J K. Multiplex gene editing in rice with simplified CRISPR- Cpf1 and CRISPR-Cas9 systems[J]., 2018, 60(8): 626-631.
[14] Li X F, Zhou W J, Ren Y K, Tian X J, Lü T X, Wang Z Y, Fang J, Chu C C, Yang J, Bu Q Y. High-efficiency breeding of early-maturing rice cultivars via CRISPR/ Cas9-mediated genome editing[J]., 2017, 44: 175-178.
[15] Zhang Q, Zhang Y, Lu M H, Chai Y P, Jiang Y Y, Zhou Y, Wang X C, Chen Q J. A novel ternary vector system united with morphogenic genes enhances CRISPR/Cas delivery in maize[J]., 2019, 181(4): 1441-1448.
[16] Al Amin N, Ahmad N, Wu N, Pu X M, Ma T, Du Y Y, Bo X X, Wang N, Sharif R, Wang P W. CRISPR-Cas9 mediated targeted disruption ofmicrosomal omega-6 desaturase in soybean (. L)[J]., 2019, 19(1): 9.
[17] 徐善斌, 郑洪亮, 刘利峰, 卜庆云, 李秀峰, 邹德堂. 利用CRISPR/Cas9技术高效创制长粒香型水稻[J]. 中国水稻科学, 2020, 34(5): 406-412.
Xu S B, Zhen H L, Liu L F, Bu Q Y, Li X F, Zou D T. Using CRISPR/Cas9 technology to efficiently create long grain fragrant rice[J]., 2020, 34(5): 406-412.(in Chinese with English abstract)
[18] 周文甲, 田晓杰, 任月坤, 魏祥进, 高扬, 谢黎虹, 刘华招, 卜庆云, 李秀峰. 利用CRISPR/Cas9创造早熟香味水稻[J]. 土壤与作物, 2017, 6(2): 146-152.
Zhou W J, Tian X J, Ren Y K, Wei X J, Gao Y, Xie L H, Liu H Z, Bu Q Y, Li X F. CRISPR/Cas9 technology was used to create early-maturing fragrant rice[J]., 2017, 6(2): 146-152.(in Chinese with English abstract)
[19] Xu Y, Lin Q P, Li X F, Wang F Q, Chen Z H, Wang J, Li W Q, Fang J, Tao Y J, Jiang Y J, Wei X D, Zhang R, Zhu Q H, Bu Q Y, Yang J, Gao C X. Fin-tuning the amylose content of rice by precise base editing of thegene[J]., 2021, 19(1): 11-13.
[20] 苏正亮. 特种稻红米品种红稻8号选育及栽培技术[J]. 乡村科技, 2020(21): 2.
Su Z L. Breeding and cultivation techniques of special rice red rice variety Hongdao 8[J]., 2020(21): 2. (in Chinese)
[21] 宗伟勋, 林建勇, 温灶婵, 梁克勤, 梁结彩, 荣建星, 邓汉儒. 特优质红米杂交水稻新组合清红优1号[J]. 杂交水稻, 2022, 37(2): 53-55.
Zong W X, Lin J Y, Wen Z C, Liang K Q, Liang C J, Ron J X, Deng H R. High quality hybrid red rice Qinghongyou 1[J]., 2022, 37(2): 53-55. (in Chinese with English abstract)
[22] 李文绍, 许鸿江, 廖荣周. 红米稻开发前景及其遗传育种研究进展[J]. 福建农业科技, 2013(4): 4
Li W Z, Xu H J, Liao R Z, Development prospect and genetic breeding of red rice[J]., 2013(4): 4. (in Chinese)
[23] Zhu Y W, Lin Y R, Chen S B, Liu H Q, Chen Z J, Fan M Y, Hu T J, Mei F T, Chen J M, Chen L, Wang F. CRISPR/Cas9 edited functional recovery of the recessiveallele to develop red rice[J]., 2019, 17(11): 2096-2105.
[24] 曾栋昌, 马兴亮, 谢先荣, 祝钦泷, 刘耀光. 植物CRISPR/Cas9多基因编辑载体构建和突变分析的操作方法[J]. 中国科学: 生命科学, 2018, 48(7): 783-794.
Zeng D C, Ma X L, Xie X R, Zhu Q L, Liu Y G. Operational methods for plant CRISPR/Cas9 multi-gene editing vector construction and mutation analysis[J]., 2018, 48(7): 783-794. (in Chinese with English abstract)
[25] Ma X L, Zhang Q Y, Zhu Q L, Liu W, Chen Y, Qiu R, Wang B, Yang Z F, Li H Y, Lin Y R, Xie Y Y, Shen R X, Chen S F, Wang Z, Chen Y L, Guo J X, Chen L T, Zhao X C, Dong Z C, Liu Y G. A robust CRISPR/Cas9 system for convenient, high-efficiency multiplex genome editing in monocot and dicot plants[J]., 2015, 8(8): 1274-1284.
[26] Hartmann L, Pedrotti L, Weiste C, Fekete A, Schierstaedt J, Gittler J, Kempa S, Krischke M, Dietrich K, Mueller M J, Vicente-Carbajosa J, Hanson J, Droge-Laser W. Crosstalk between two bZIP signaling pathways orchestrates salt-induced metabolic reprogramming inroots[J]., 2015, 27: 2244-2260.
[27] Fukuda A, Nakamura A, Hara N, Toki S, Tanaka Y. Molecular and functional analyses of rice NHX-type Na+/H+antiporter genes[J]., 2011, 233(1): 175-188.
[28] Passricha N, Saifi S K, Kharb P, Tuteja N. Rice lectin receptor-like kinase provides salinity tolerance by ion homeostasis[J]., 2020, 117(2): 498-510.
[29] Ogo Y, Itai R N, Nakanishi H, Kobayashi T, Takahashi M, Mori S, Nishizawa N K. The rice bHLH protein OsIRO2 is an essential regulator of the genes involved in Fe uptake under Fe-deficient conditions[J]., 2010, 51(3): 366-377.
[30] Kobayashi T, Ogo Y, Itai R N, Nakanishi H, Takahashi M, Mori S, Nishizawa N K. The transcription factor IDEF1 regulates the response to and tolerance of iron deficiency in plants[J]., 2007, 104(48): 19150-19155.
[31] Tan S, Yin L P. Research of the iron-regulated transporter 1 (IRT1) in the past decades and its latest development[J]., 2017, 62(5): 350-359.
Breeding ofFunction Restoration Red Rice via CRISPR/Cas9 Mediated Genome Editing
ZHANG Yuanye1, YIN Liying1, LI Rongtian1,*, HE Mingliang2, LIU Xinxin3, PAN Tingting4, TIAN Xiaojie2, BU Qingyun2, LI Xiufeng2,*
(1College of Life Science, Heilongjiang University, Harbin 150080, China;2Northeast Institute of Geography and Agroecology, Key Laboratory of Soybean Molecular Design Breeding, Chinese Academy of Sciences, Harbin 150081, China;3College of Life Science, Northeast Forestry University, Harbin 150040, China;4College of Agriculture, Heilongjiang Bayi Agricultural University, Daqing 163319, China;*Corresponding author, email: lirongtian07@aliyun.com; lixiufeng@iga.ac.cn)
【Objective】It is of great value to restore cultivated rice varieties to red rice with good rice quality and strong stress resistance. CRISPR/Cas9 gene editing technology was used to edit thegene, restoring the red seed coat, and laying a data foundation for the improvement of rice quality and resistance.【Methods】Using CRISPR/Cas9 technology, the target genemutant vector pYLCRISPR/ Cas9--gRNA was constructed and transformed into transgenic plants with Kongyu 180 and Shangyu 453 as materials. The results were verified by sequencing and phenotypic observation. 【Results】 Twomutants were obtained. For KY-1, with 4 bases deleted from 1414bp to1417 bp, the terminator was replaced by phenylalanine. SY-1 also lost a base at 1411 bp, which resulted in the transformation from the terminator to aspartic acid. The two kinds of editing materials restored red rice phenotype and had certain saline-alkali tolerance.【Conclusion】Using CRISPR/Cas9 technique, we obtained homozygous lines with red seed coat, providing basic materials for the improvement of red rice.
rice;; red episperm; salinity tolerance; CRISPR/Cas9
10.16819/j.1001-7216.2022.211205
2021-12-07;
2022-03-10。
黑龙江省杰出青年基金资助项目(JQ2020C003); 中国科学院战略性先导科技专项(A类)(XDA28100000)。
猜你喜欢 红米种皮株系 东北地理所发现PG 031基因具有改良大豆种皮吸水性的应用潜力粮油与饲料科技(2022年2期)2022-11-24过表达NtMYB4a基因增强烟草抗旱能力亚热带植物科学(2022年1期)2022-05-17花生种皮颜色智能识别模型的建立与应用中国油料作物学报(2022年2期)2022-05-13盘州市红米种植的气候适应性探究南方农业(2021年32期)2022-01-14菜豆种皮开裂过程中木质素含量及相关酶活变化黑龙江大学工程学报(2021年2期)2021-06-22红米Redmi Note 7电脑报(2019年8期)2019-09-10红米怎么吃农产品市场周刊(2019年3期)2019-07-29红米S2:自拍最好手机通信产业报(2018年16期)2018-08-20衢州椪柑变异株系—黄皮椪柑相关特性研究浙江柑橘(2016年1期)2016-03-11脱脂椰子种皮多肽的抗氧化活性研究中国粮油学报(2014年8期)2014-02-06







